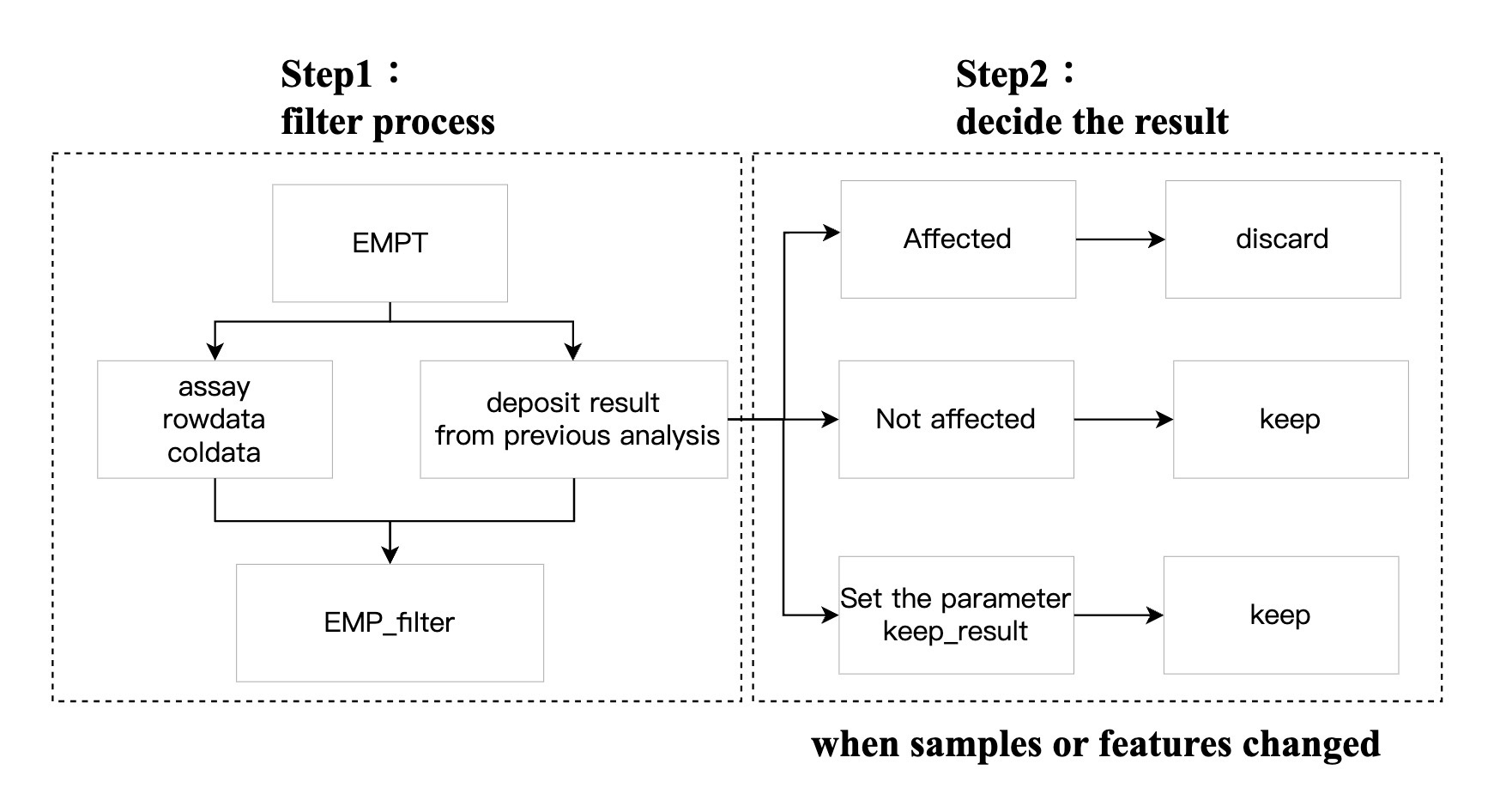
7.1 数据筛选 EMP_filter
模块EMP_filter功能十分强大,不仅可以根据数据的rowdata和coldata对特征和样本进行筛选、过滤,还可以对数据分析结果进行筛选。为了便于用户理解,以下对模块EMP_filter的基本参数进行介绍:
- obj : 指定待分析的对象MAE或者EMPT。
- experiment : 指定待分析组学项目的名称(character)。
- sample_condition:设定选择样本的阈值条件。
- feature_condition: 设定选择特征的阈值条件。
- filterSample: 直接指定要筛选的样本名称。
- filterFeature: 直接指定要筛选的特征名称。
- action: 与
filterSample和filterFeature联用,设定为kick或select。 - keep_result: 当输入值为 TRUE 时,系统将保留所有分析结果,样本与特征的任何更改均不影响此设置。当输入值为某个名称时,则仅该名称所对应的分析结果会被予以保留。
- show_info: 设定输出结果的展现方式。
①用户需理解模块
EMP_filter的过滤逻辑,首先是根据参数sample_condition和feature_condition的指定条件筛选样本和特征,然后再根据参数filterSample和filterFeature选择或者剔除样本和特征。②如果一次
EMP_filter无法满足筛选需求,可以多次进行EMP_filter筛选过程。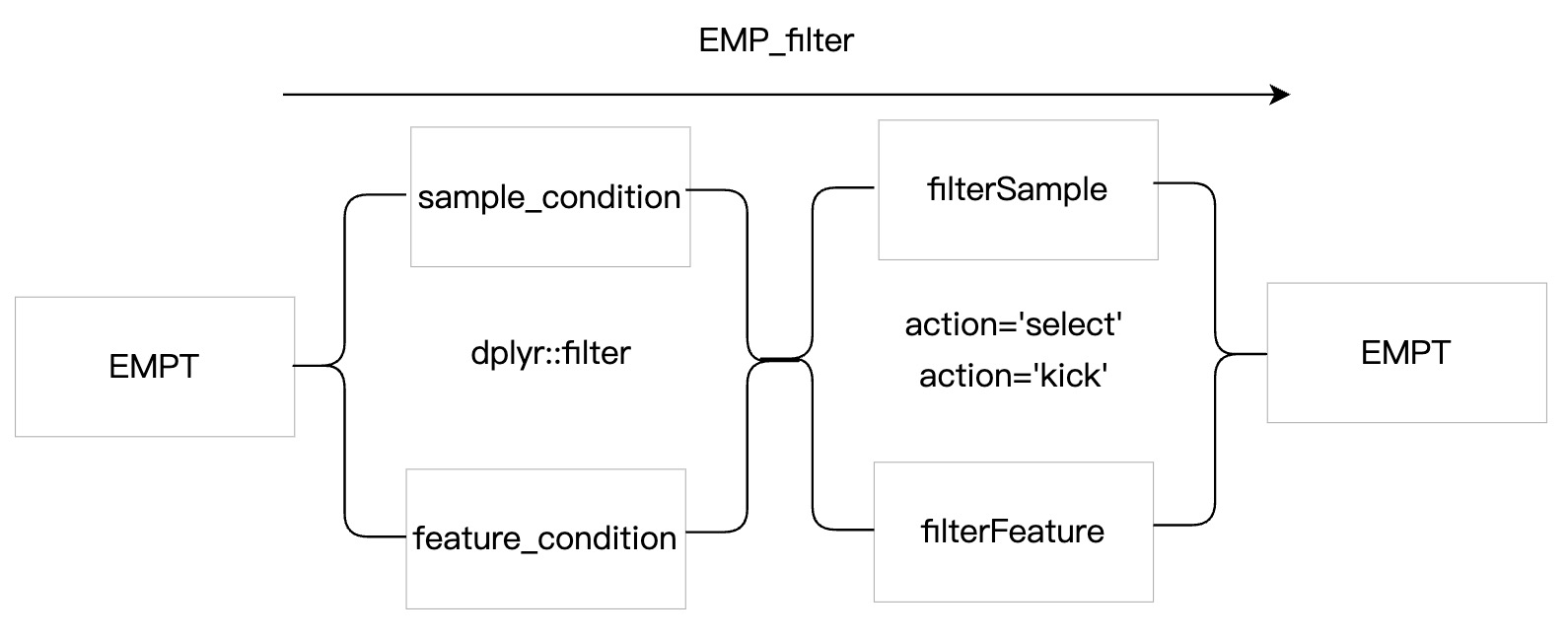
7.1.1 对所有组学项目数据进行筛选过滤
🏷️示例1:在所有组学项目中筛选年龄大于30岁的男性受试者。
MAE |>
EMP_filter(sample_condition = Sex == 'M' & Age >30)
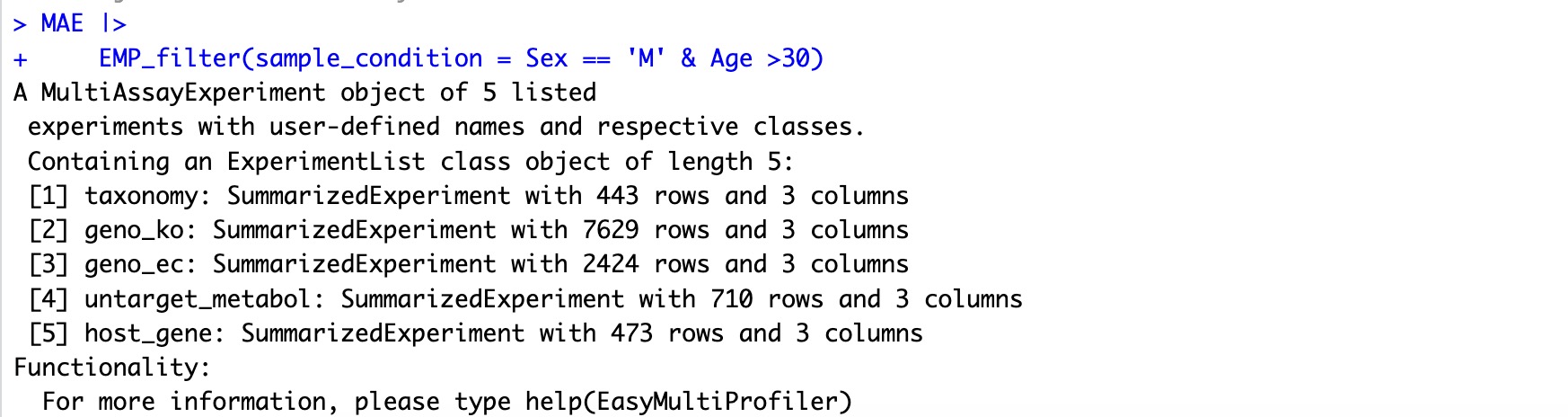
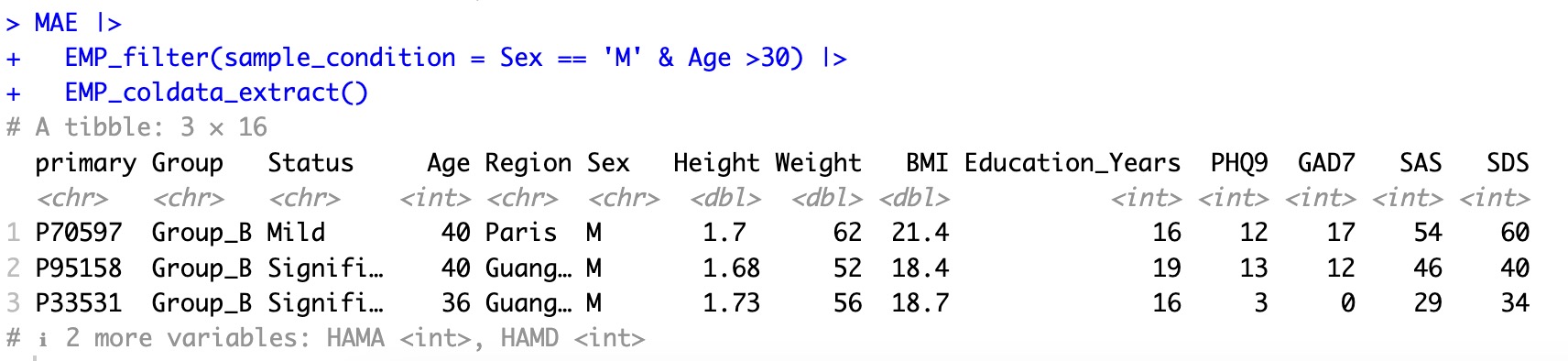
筛选完成后,MAE对象中只存在满足条件的样本,可以直接使用其他分析模块进行下游分析。例如可以使用模块EMP_coldata_extract观察过滤后样本情况。
MAE |>
EMP_filter(sample_condition = Sex == 'M' & Age >30) |>
EMP_coldata_extract()
🏷️示例 2:筛选在表型数据中没有缺失值的样本
模块
EMP_filter继承了dplyr包的filter模块语法,因此可以使用tidy语法进行样本筛选。
MAE |>
EMP_filter(sample_condition = if_all(everything(),~ !is.na(.)))
🏷️示例3:筛选出PHQ9和GAD7至少有一个取值大于5的样本。
MAE |>
EMP_filter(sample_condition = if_any(c(PHQ9,GAD7),~. > 5))
7.1.2 对单一组学项目数据及分析结果进行筛选过滤
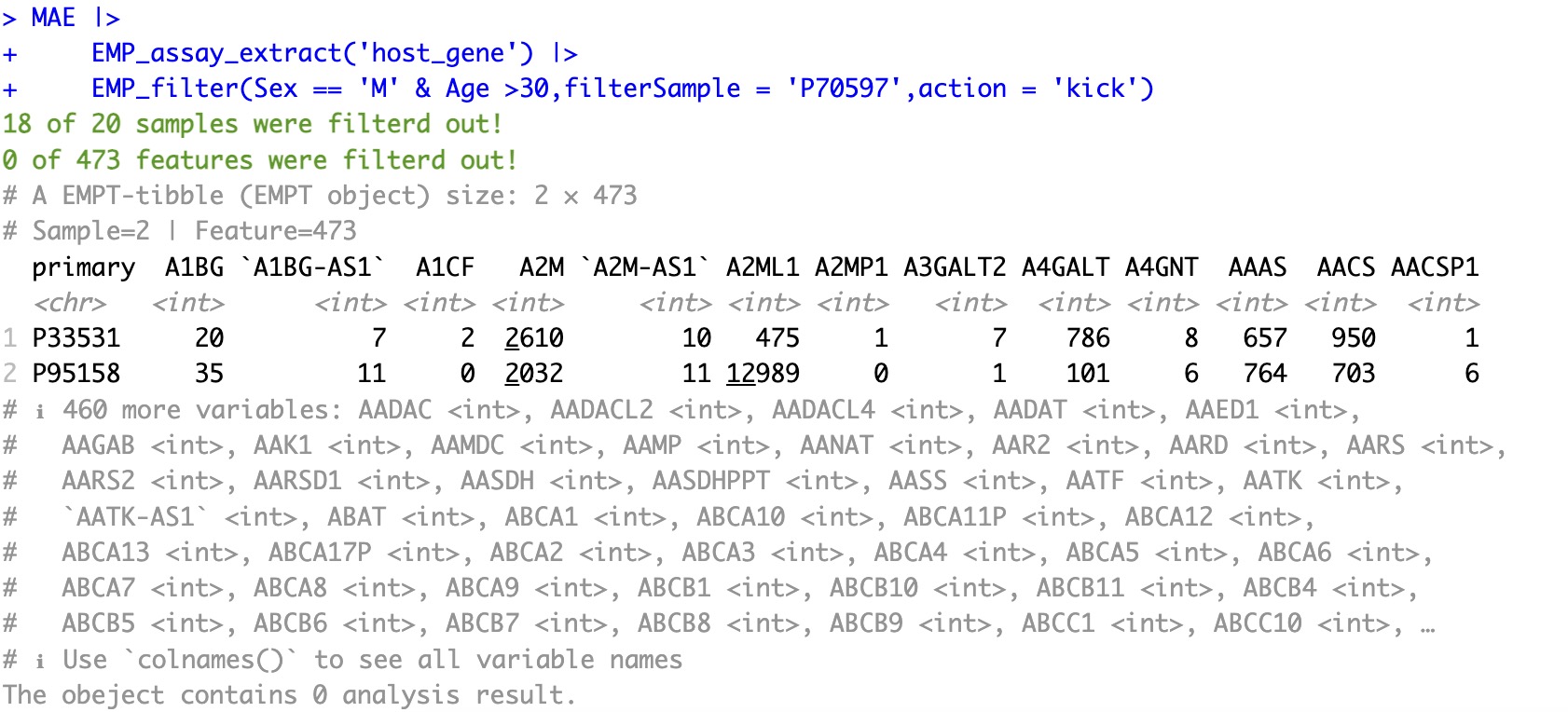
🏷️示例1:根据原始数据筛选样本。
参数
action仅与参数filterSample和filterFeature配合使用。从MAE对象中提取组学项目host_gene的assay,筛选出sex为M的样本,并将受试者编号为P70597的样本剔除。
MAE |>
EMP_assay_extract('host_gene') |>
EMP_filter(Sex == 'M' & Age >30,filterSample = 'P70597',action = 'kick')
🏷️示例2:根据数据分析结果筛选样本。
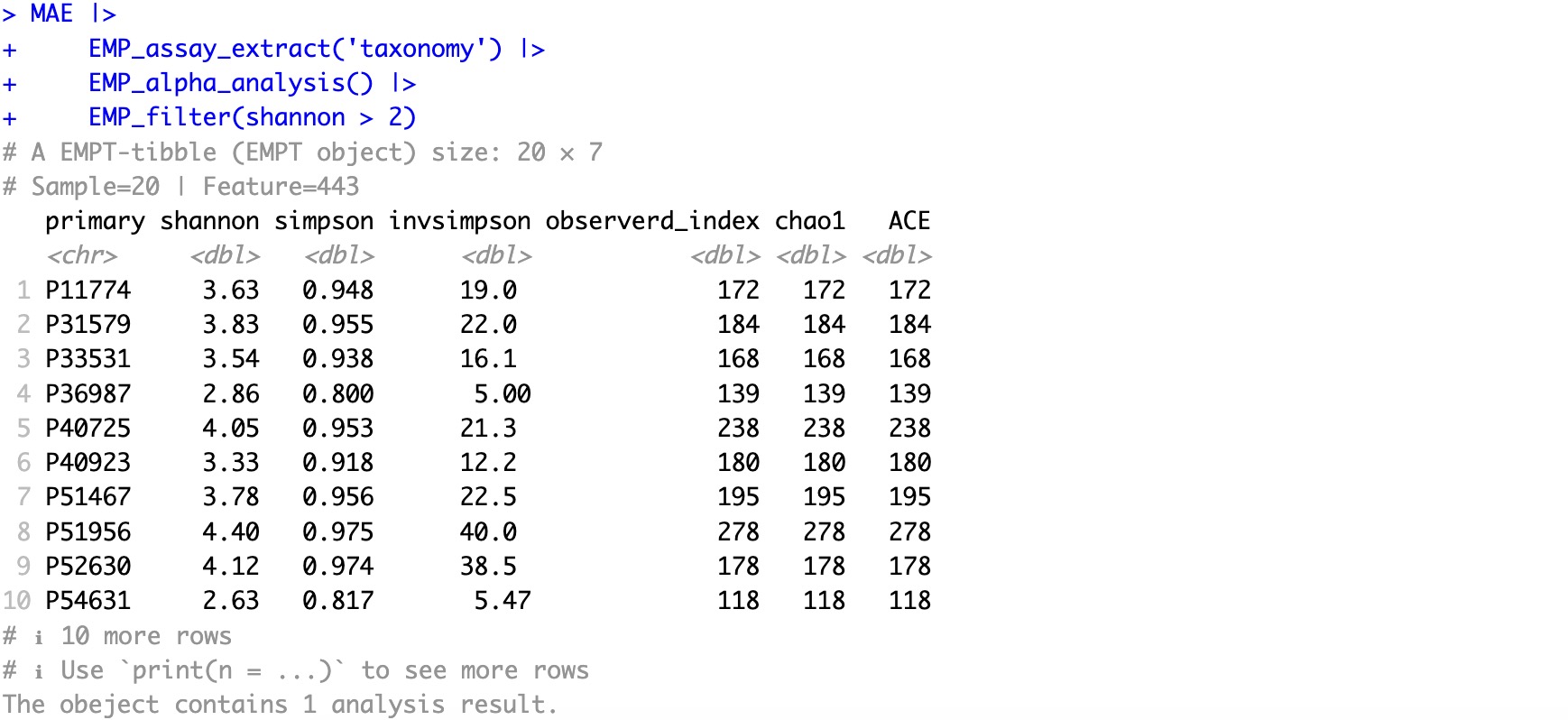
从MAE对象中提取taxonomy的assay,进行alpha多样性分析,筛选满足shannon大于2的样本。
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_alpha_analysis() |>
EMP_filter(shannon > 2)
🏷️示例3:根据数据分析结果筛选特征。
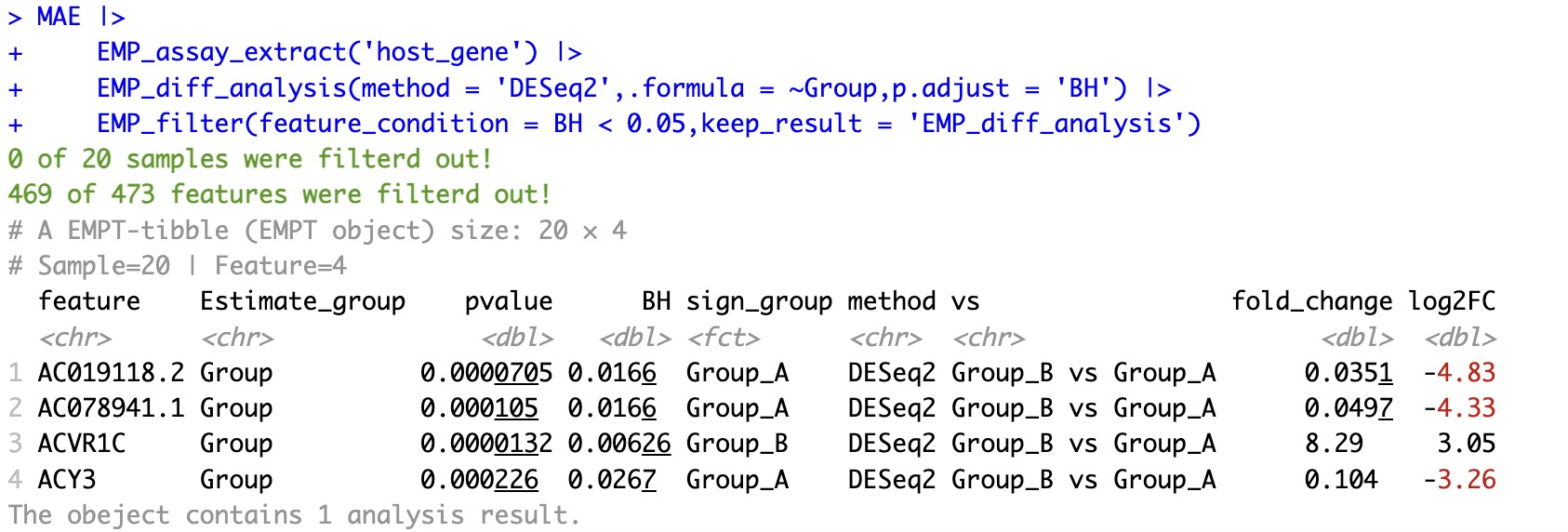
从MAE对象中提取组学项目host_gene的assay,进行差异性检验,并筛选出校正p值小于0.05的特征。
①由于在DESeq2算法中,变更特征和样本后的差异分析结果均会发生改变,因此如果需要原有差异分析结果需要参数
keep_result。②传统的统计检验,如t.test或者wilcox.test等,不受特征改变,因此无需参数
keep_result即可保留此结果。
MAE |>
EMP_assay_extract('host_gene') |>
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group,p.adjust = 'BH') |>
EMP_filter(feature_condition = BH < 0.05,keep_result = 'EMP_diff_analysis')
🏷️示例4:多次、多条件筛选样本或特征。
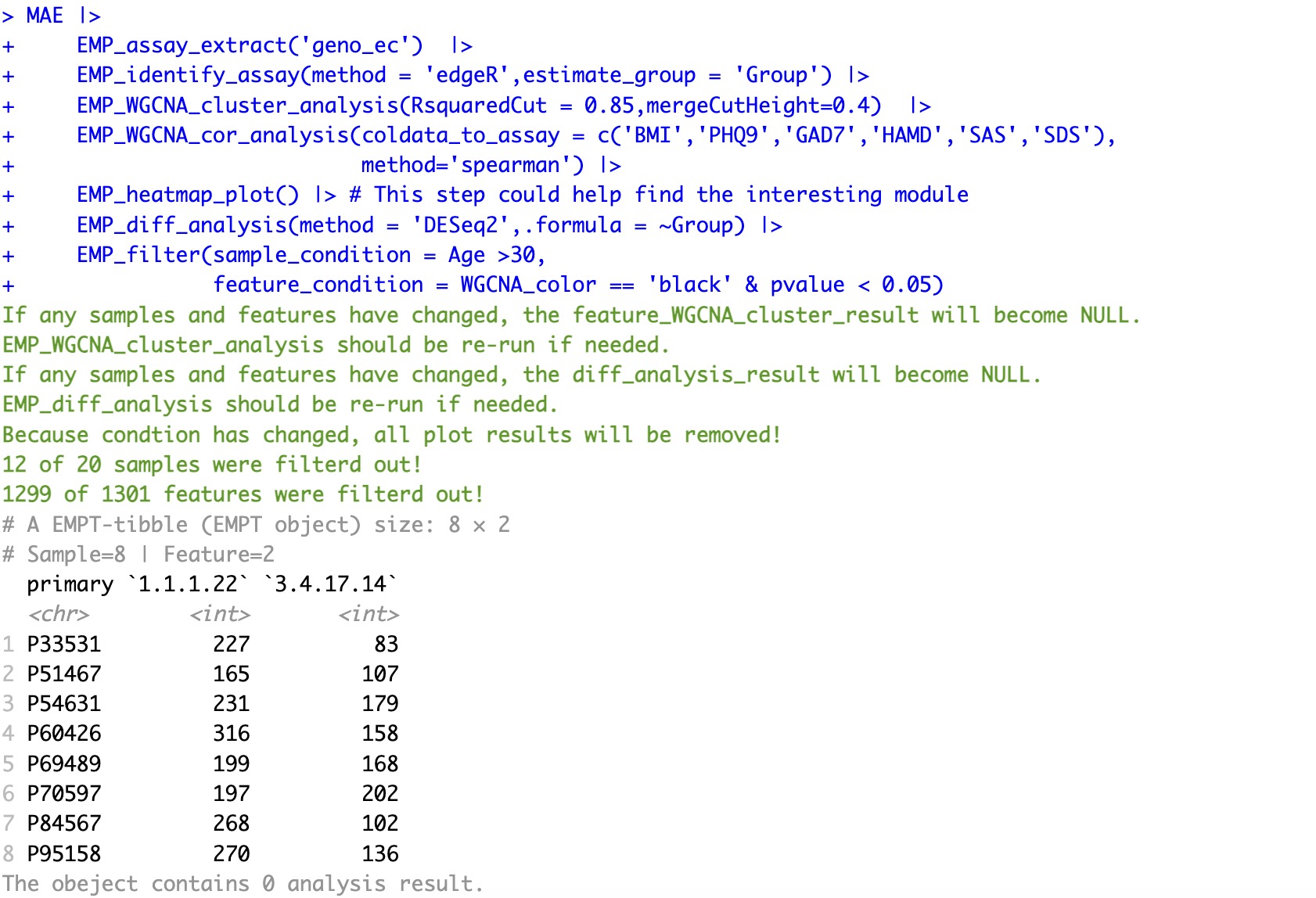
从MAE对象中提取组学项目geno_ec的assay,进行WGCNA关联分析后再进行DESeq2差异性分析, 最后筛选出以下条件的数据:
- 年龄大于30岁的样本;
- 使用edgeR筛选核心基因;
- 在WGCNA关联分析中,与BMI的相关性具有统计学意义的black模块聚类内的特征;
- 差异性分析中p值小于0.05的特征。
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_identify_assay(method = 'edgeR',estimate_group = 'Group') |>
EMP_WGCNA_cluster_analysis(RsquaredCut = 0.85,mergeCutHeight=0.4) |>
EMP_WGCNA_cor_analysis(coldata_to_assay = c('BMI','PHQ9','GAD7','HAMD','SAS','SDS'),
method='spearman') |>
EMP_heatmap_plot() |> # This step could help find the interesting module
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group) |>
EMP_filter(sample_condition = Age >30,
feature_condition = WGCNA_color == 'black' & pvalue < 0.05)
7.1.3 关于过滤后数据分析结果的清除问题
在EasyMultiProfiler包的分析流程中,分析结果会自动存储在对象中,供模块EMP_filter用于筛选样本和特征。然而,一旦筛选完成,之前存储的结果可能不再适用,因此会在筛选后自动清除(例如:容器中存储了差异性分析和alpha多样性分析的结果,然后用户根据BMI值剔除了一些样本。在这种情况下,原有的差异性分析结果将会自动清除,因为它们已经不再有效;而alpha多样性的结果则不受剔除样本的影响,会继续保存在容器中,以便下一次筛选使用)。EasyMultiProfiler包已经实现了自动化处理这一过程,并且会以红色文字提醒用户已经执行了该操作。
容易误解的例子:
🏷️示例 : 以EC:1.1.1.1的差异分析为例子。
当对全部特征进行deseq2差异分析如下:
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group)
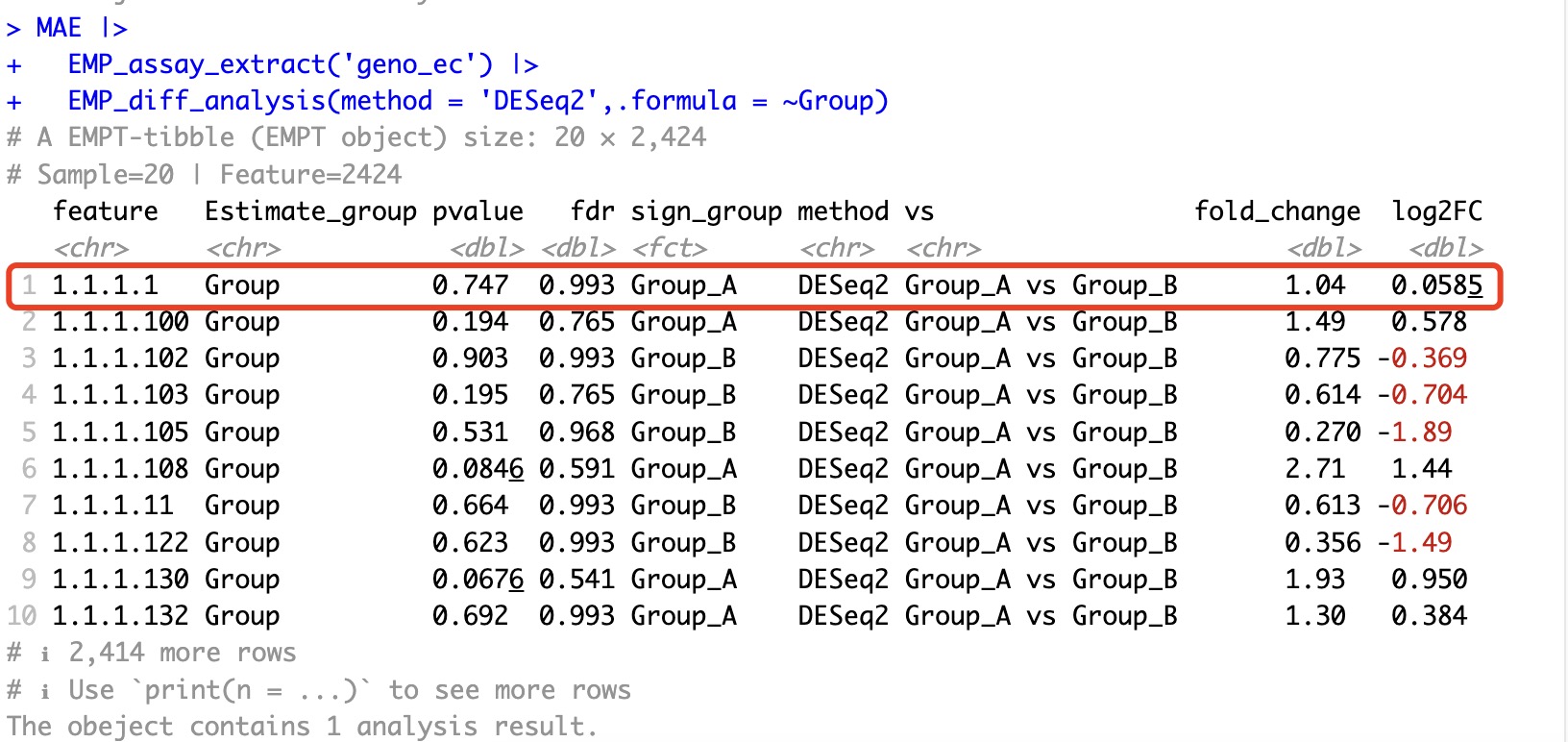
当只剔除部分其他特征后,再次进行DESeq2差异分析,可以发现1.1.1.1的结果发生了改变。这是因为DESeq2算法本身原因所致。这种情况还出现在edgeR、limma等算法中。
MAE |>
EMP_assay_extract('geno_ec',pattern = '1.1.1.1',pattern_ref = 'feature') |>
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group)
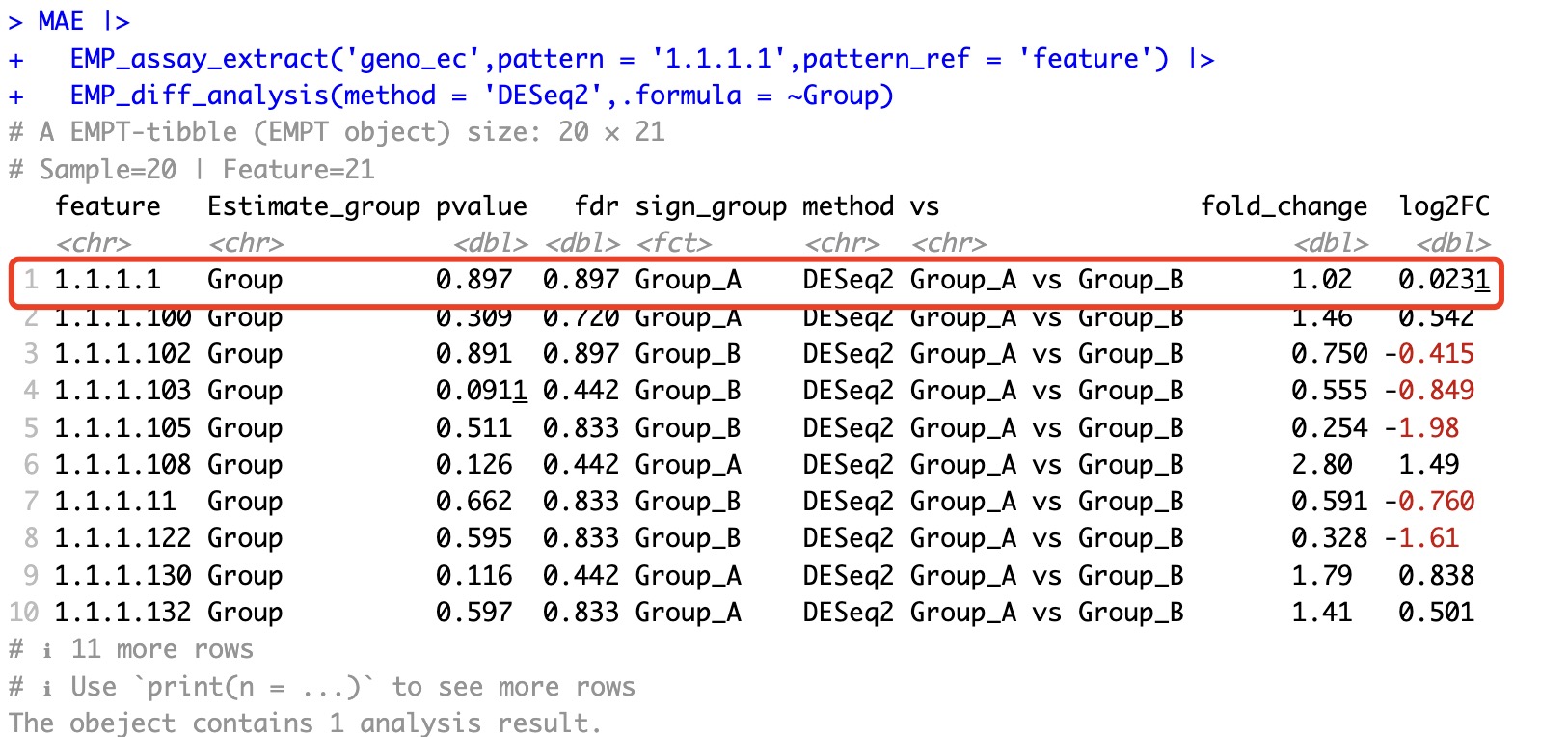
但是,在传统的统计检验中,则不会出现类似的情况。
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_diff_analysis(method = 't.test',estimate_group = 'Group')
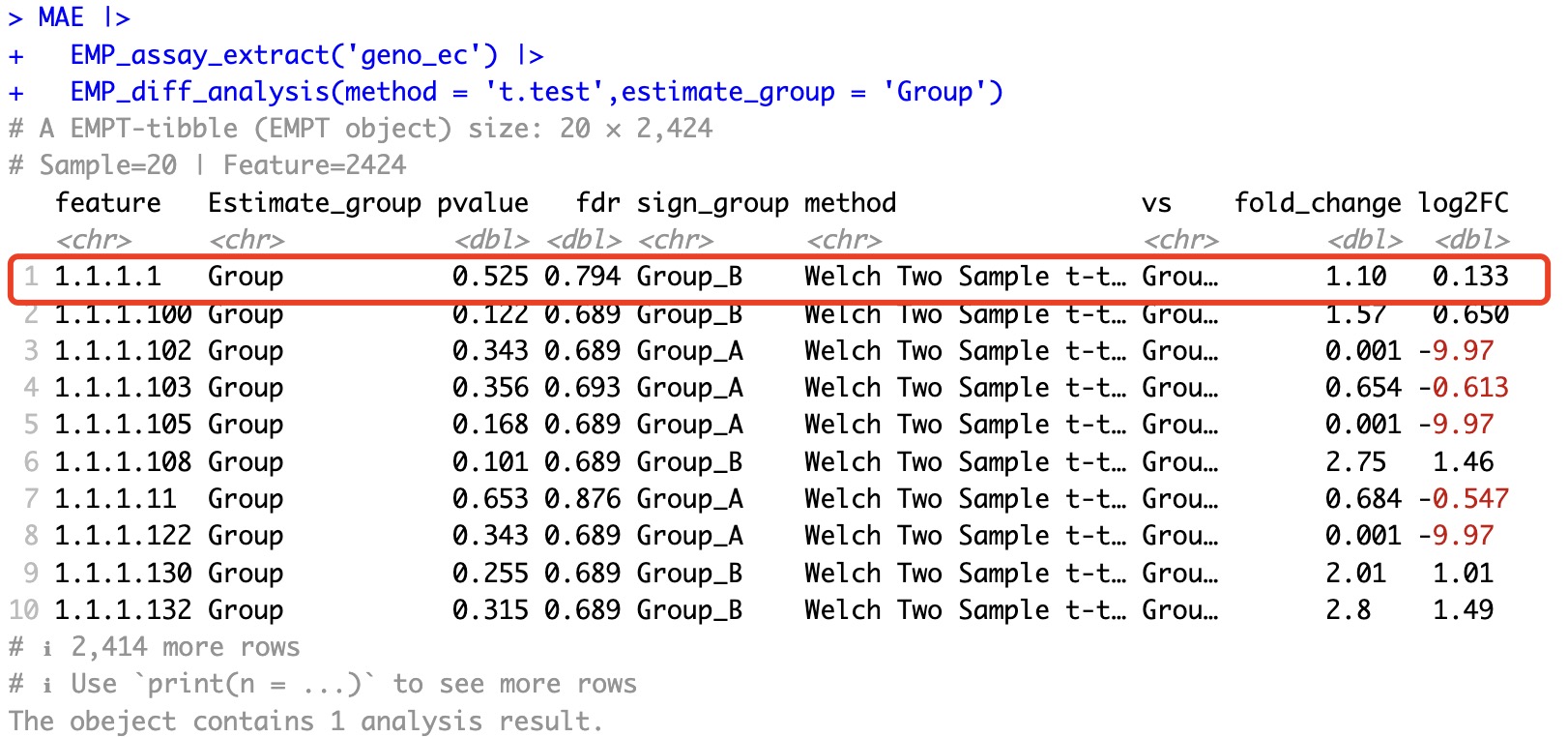
MAE |>
EMP_assay_extract('geno_ec',pattern = '1.1.1.1',pattern_ref = 'feature') |>
EMP_diff_analysis(method = 't.test',estimate_group = 'Group')
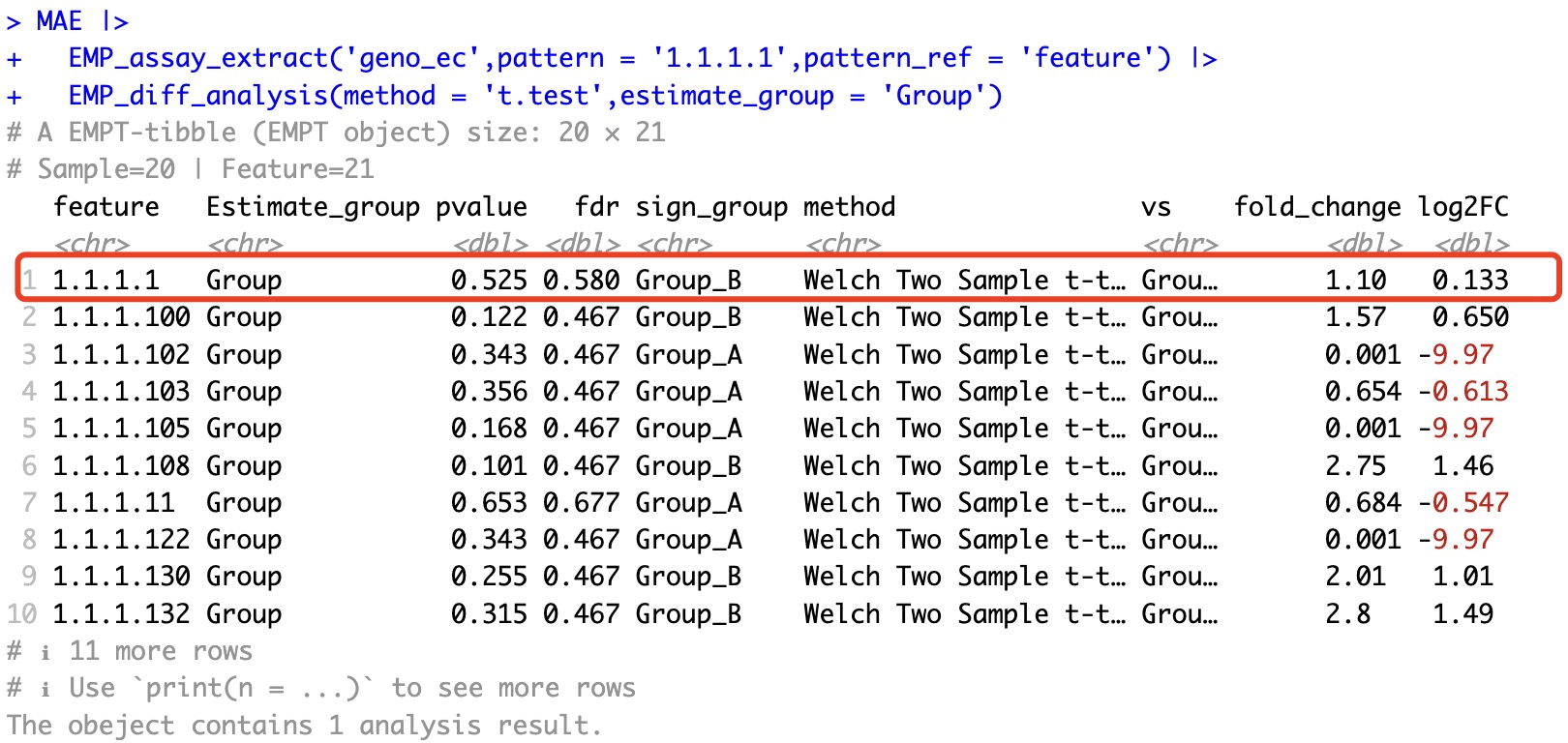
因此,当特征数目发生改变时候,EMP_filter会保留传统统计的差异结果,而清除DESeq2、edgeR和limma算法的结果。例如我们在DESeq2差异分析后筛选p值小于0.05的特征,由于特征数目减少,因此会触发清除动作。由于在完成筛选后,差异分析结果被清除后,因此输出结果为assay矩阵。
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group) |>
EMP_filter(feature_condition = pvalue < 0.05)
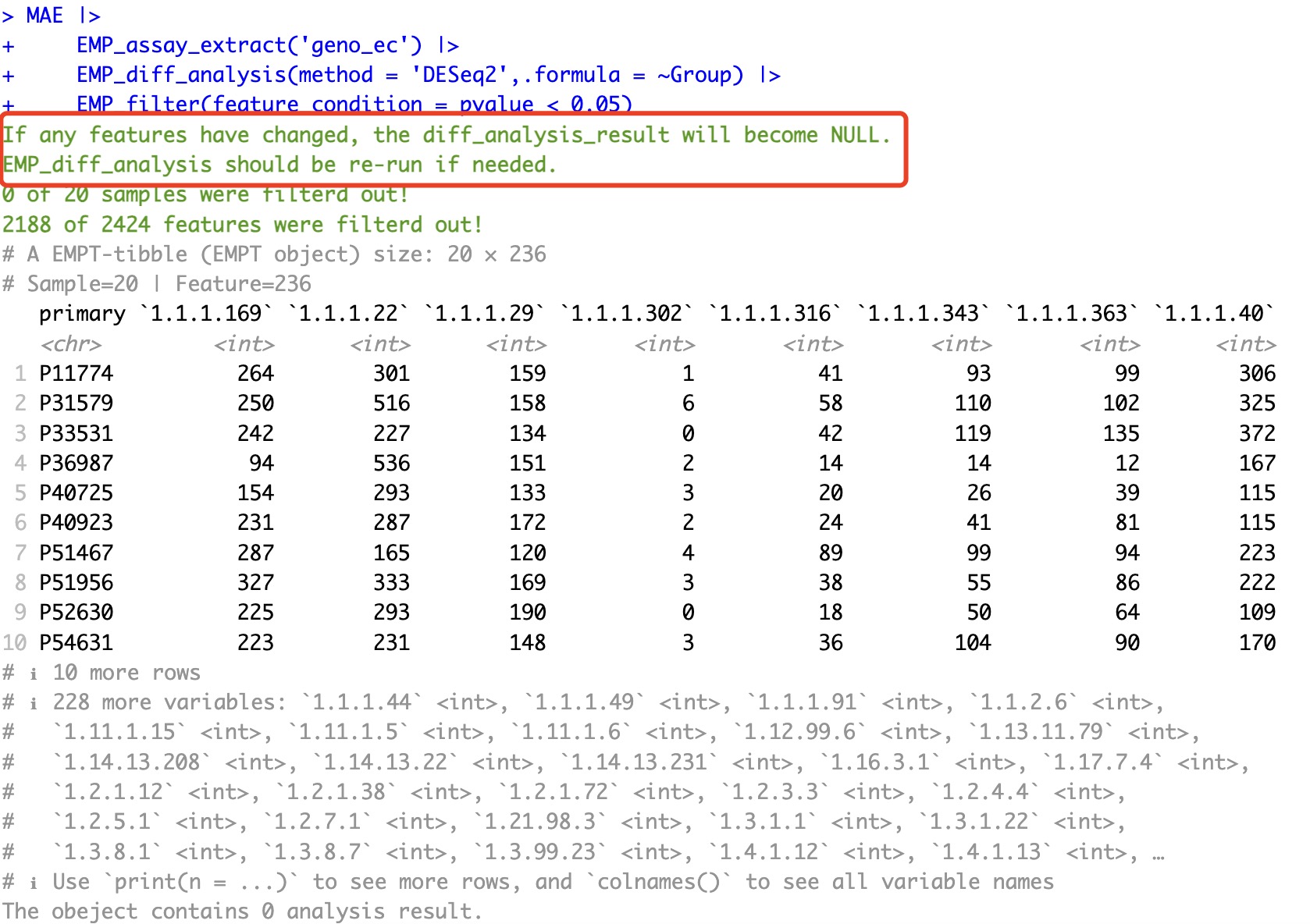
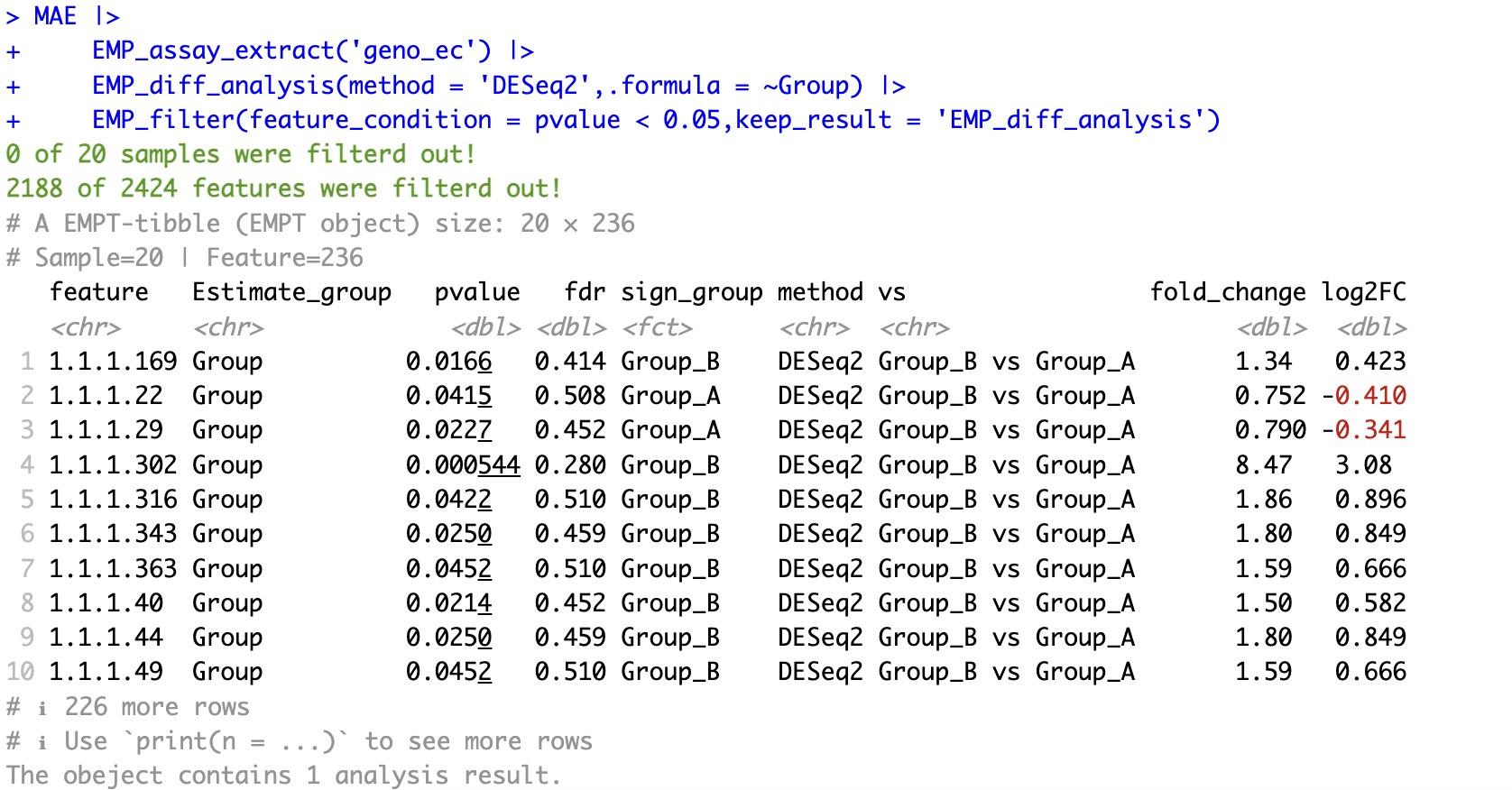
如果,坚持要保留相应的结果,可以使用keep_result参数保留相应分析的结果。
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group) |>
EMP_filter(feature_condition = pvalue < 0.05,keep_result = 'EMP_diff_analysis')
7.1.4 关于过滤后数据的显示问题
在EasyMultiProfiler包的分析流程中,使用模块EMP_filter筛选后,默认会继承前一个分析模块的显示效果(例如:如果前一个模块是进行了alpha多样性计算,当使用模块EMP_filter根据Age>30条件进行筛选时,由于样本的变化不会影响到原先的alpha多样性结果,因此模块EMP_filter的输出将会继续显示上一个模块中经过筛选的结果,即模块EMP_alpha_analysis的筛选结果)。如果前一个模块因为样本或特征的改变而被清除(详见7.1.3的说明),则会直接显示当前数据集assay的情况。
7.1.5 关于过滤后数据提取问题
在模块EMP_filter中,参数action仅用于配合filterSample和filterFeature进行筛选使用。如果需要在筛选后提取assay数据矩阵,可以在模块EMP_filter后继续使用模块EMP_assay_extract并指定参数action='get'。
🏷️示例:筛选后提取assay数据矩阵。
MAE |>
EMP_assay_extract('host_gene') |>
EMP_filter(Sex == 'M' & Age >30,filterSample = 'P70597',action = 'kick') |>
EMP_assay_extract(action = 'get')
7.1.6 关于过滤时变量引入的问题
由于模块EMP_filter继承了dplyr包的filter模块语法,因此当你想从 env 变量中获取数据变量而不是直接键入数据变量的名称时, 应当使用语法。
请务必遵循这种间接引入的正确用法,否则可能会出现错误的过滤结果。
🏷️示例:
target_status = c("Mild","No")
MAE |>
EMP_filter(Status %in% target_status) ## Wrong usage!!
MAE |>
EMP_filter(Status %in% {{target_status}}) ## Correct usage!!